| El virus
causante de la Fiebre Aftosa fue el primer "agente filtrable"
responsabilizado de causar enfermedad en los animales por Löefler
y Frosch a finales del siglo XIX. Desde entonces, se
han sucedido en la literatura científica un gran número
de investigaciones y hallazgos en relación con este virus.
De hecho, el virus de la Fiebre Aftosa
constituye uno de los patógenos que afectan a los animales
domésticos mejor y más profundamente conocidos.
|
El virus de la Fiebre Aftosa
es un Aphtovirus de la familia Picornaviridae.
Se trata, por tanto, de un virus ARN monocatenario cuyo
genoma se encuentra incluido en una cápside proteica
de morfología icosaédrica formada por protómeros
integrados por 4 proteínas estructurales
distintas que reciben las designaciones VP1, VP2, VP3 y
VP4. De entre ellas, la proteína
VP1 es la más importante, ya que constituye
uno de los antígenos más inmunógenos
y biológicamente activos del virus, al intervenir
en el reconocimiento de los receptores celulares y la formación
de anticuerpos neutralizantes. Esta proteína es,
además, altamente variable, lo que la hace responsable
en gran medida de esta característica del virus.
|
|
|
|
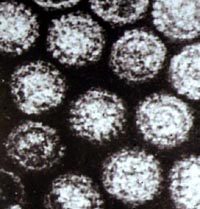
Partículas del virus de
la fiebre aftosa por microscopía electrónica
de transmisión.
|
| El genoma vírico, integrado
por ARN monocatenario de polaridad
positiva (de unos 8500 nucleótidos), se estructura en
una fase de lectura abierta (Open Reading Frame, ORF) central
situada entre dos zonas no codificadoras. De ellas, la zona
no codificadora situada en el extremo 5' presenta unida covalentemente
una proteína (VPg) que participa en el inicio de la neosíntesis
de ARN vírico. Existe además otra región
en el genoma vírico que determina una estructura muy
conservada, denominada IRES, e implicada en el reconocimiento
de ribosomas en el interior de la célula hospedadora. |
|
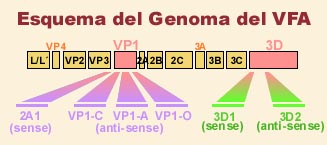
Esquema del genoma del virus de la Fiebre aftosa
|
|
La zona ORF codifica tres
poliproteínas (P1-2A, P2 y P3) que, posteriormente,
sufrirán diversos cortes enzimáticos por proteasas
víricas. El precursor P1-2A originará finalmente
las cuatro proteínas estructurales antes mencionadas,
mientras que P2 y P3 darán lugar a otras 8 proteínas
no estructurales. Estas, junto con diversos precursores,
realizan funciones necesarias para la compleción
del ciclo vírico intracelular: las derivadas de P2
(2A, B y C) intervienen en la inducción de síntesis
de ARN vírico o en la formación de vesículas
en membrana; por su parte, las originadas a partir de P3
(3ABC y D) intervienen en el anclaje a células hospedadoras,
el efecto citopático, la inhibición de síntesis
proteica celular y, especialmente relevante es la acción
ARN Polimerasa de 3D, la antiguamente conocida como antígeno
vírico asociado a infección (VIA).
|
|
Las proteínas estructurales se
ensamblan en pentámeros hasta formar, junto
a la incorporación de ARN vírico de novo, nuevas
partículas víricas. Estas responden a la estructura
genérica de los Picornaviridae, esto es, carecen
de envoltura, presentan simetría
icosaédrica y un tamaño en torno a los 30 nm
de diámetro.
Desde el punto de vista estructural, una de las características
más relevantes del virus de la Fiebre Aftosa es la
disposición de la proteína VP1. Esta, junto
con VP2 y VP3 ocupa posiciones externas y es por tanto activa
en fenómenos de reconocimiento y antigenicidad. En
el caso de VP1, además, destaca el hecho de que parte
de su cadena aminoacídica se dispone en forma de bucle
proyectado hacia el exterior presentando una gran movilidad,
funcionalidad y variabilidad. Es, por ejemplo, esencial
en el anclaje a células hospedadoras, constituye un
punto básico en reacciones de neutralización
y puede cambiar con gran facilidad su conformación
espacial sin que el resto de la partícula vírica
sufra modificación alguna.
Las diferentes secuencias genómicas
que codifican VP1 son precisamente las responsables de la
existencia de los diferentes serotipos y subtipos que,
tradicionalmente, han sido establecidos en base a reacciones
de base humoral.
|
Inmunológicamente
pueden distinguirse 7 tipos, que responden a las
denominaciones A (Allemagne),
O (Oise), C
(así denominado asumiendo que los anteriores serían
renombrados "A" y "B"), SAT1,
SAT2, SAT3
(por South African Territories) y Asia1.
Dentro de cada serotipo existen numerosos
subtipos, hasta un total de aproximadamente 65. El
agrupamiento en estas categorías, inicialmente se
basaba en estudios de protección cruzada en animales,
y luego, a efectos de su estandarización, fue establecida
mediante paneles de sueros.
|
|
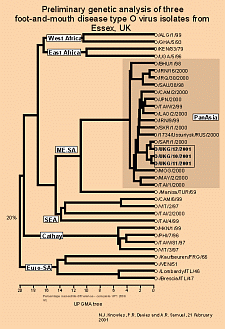
Árbol filogenético del virus inductor del brote
de Fiebre aftosa del reino Unido del año 2001.
Pulse sobre la imagen para verla a tamaño real.
|
|

Inmunológicamente se han descrito 7 tipos 65 subtipos
del virus de la Fiebre aftosa.
|
|
El
análisis de las secuencias de ARN que codifican VP1 en
los diferentes serotipos ha permitido establecer las relaciones
filogenéticas entre los distintos aislados que,
en términos generales, se corresponden con las divisiones
inmunológicas tradicionales, con dos grupos bien definidos:
los que constituyen los serotipos SAT de una parte, y los A,
O, C y Asia 1 por otra. |
|
A la gran diversidad de
serotipos y subtipos debe añadirse la circunstancia
de que el virus de la Fiebre Aftosa es extraordinariamente
variable desde el punto de vista
genómico, con una gran facilidad para modificar las
secuencias que codifican tanto sus proteínas estructurales
como no estructurales, así como de hacer estables las
nuevas variaciones.
Se han descrito mecanismos
para este fenómeno similares a los que presentan los
virus influenza (entre los que se cuenta el de la gripe),
el denominado drift antigénico, esto es, un
cambio menor caracterizado por una cierta cantidad de reemplazos
aminoacídicos. Pero, igualmente que este tipo de modificaciones,
se han descrito otros cambios de gran
magnitud en los que ciertos aminoácidos de localización
crítica se ven sustituidos, alterándose de esta
forma varios epitopos o determinantes antigénicos a
un tiempo (lo que se conoce para los virus influenza
precisamente como shift antigénico). Incluso
pueden darse variaciones antigénicas en el curso de
una misma onda epizoótica. La variabilidad mencionada
no sólo se restringe a elementos estructurales o incluso
funcionales, sino que pueden determinar una marcada modificación
de las propiedades biológicas, entre ellas la virulencia
del virus para diversas especies animales.
|
 Imprimir
Imprimir
|